疾病与健康
动物
植物
微生物
数据库
工具
Nat Genet背靠背成果专辑数据库上线,描绘肝脏区域性代谢、损伤和再生高分辨率时空组学图谱
2024-04-22 149CNGBdb
2024年4月16日,国家基因库生命大数据平台支撑科研成果在《Nature Genetics》背靠背发表。中国科学院分子细胞科学卓越创新中心惠利健研究组与杭州华大生命科学研究院研究团队,联合吉林大学、广州医科大学第五附属医院、山西医科大学、上海科技大学、IRCCS-Istituto Tumori、汉诺威医学院等国内外机构,利用Stereo-seq和DNBelab C4技术,分别揭示了小鼠肝脏在胆汁淤积损伤与修复过程,以及在肝脏稳态、切除再生中的复杂分子机制。两项研究不仅构建了一个全面的肝脏参考图谱,可用于发现区域性肝脏损伤再生中的潜在机制和靶点,也为研究其它具有异质性分布的组织在损伤与再生过程中的时空细胞动态、分子机制提供了新的研究范式。

同时,上述两项研究的专辑数据库已于国家基因库生命大数据平台(CNGBdb)同步上线,开放相关资源给全球科研同行使用。此外,上述两项研究的相关数据均存储于国家基因库生命大数据平台(CNGBdb),项目编号分别为:CNP0003447 和CNP0002310。
胆汁淤积与再生时空转录图谱
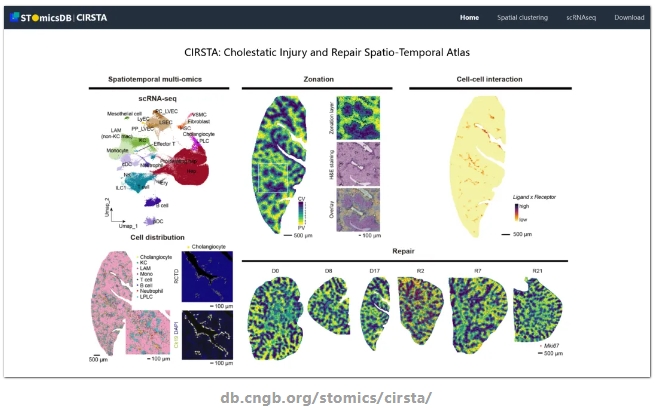
中国科学院分子细胞科学卓越创新中心惠利健研究组联合杭州华大生命科学研究院,在线发表了题为A spatiotemporal atlas of cholestatic injury and repair in mice的研究论文。该研究以细胞水平的空间分辨率描绘了胆汁淤积与再生过程中的损伤响应和微环境信号的时空动态变化特征,发现胆管细胞在胆汁淤积损伤下作为信号枢纽,与门静脉区域免疫细胞重塑和肝细胞重编程高度相关。同时,研究还发现Atoh8是限制肝细胞增殖的重要因子,为解析肝脏区域性损伤以及胆管相关疾病的治疗提供了新的视角和可能的新靶点。
胆汁性淤积损伤后,胆管细胞大量增殖,可能影响门静脉区的损伤,但具体细节尚待探究;门静脉区肝细胞会重编程成类肝前体细胞(Liver progenitor-like cell,LPLC),贡献肝脏再生。但重编程产生的LPLC只有一部分会贡献到胆管细胞,因此LPLC及其微环境可能存在异质性;随着损伤撤去后,肝脏修复起始,肝细胞大量增殖,这一现象主要发生在中央静脉区。这些损伤再生模式与其空间位置高度相关,所以在空间维度进行解析是非常重要的。
通过对胆汁淤积损伤下门静脉区域的空间互作进行深入分析,研究人员发现胆管可能充当信号枢纽,整合门静脉区域环境中的细胞互作。数据显示胆管细胞分泌的Csf1与单核细胞到脂质相关巨噬细胞(LAM)的分化相关。同时,研究揭示了肝细胞重编程过程中LPLC的两种亚型。胆管细胞通过分泌转化生长因子Tgfβ,与LPLC2向胆管属性转变高度相关。
在损伤的过程中,研究人员发现了强烈的促增殖信号,然而肝细胞并未有明显的增殖,这提示损伤过程中可能存在增殖抑制因子。通过转录因子调控网络分析,Atoh8被识别为一个可能的抑制因子。进一步实验验证表明,敲降Atoh8可以显著促进肝脏再生。
综上,这项研究不仅构建了胆汁淤积损伤与再生的时空转录组图谱,还强调了胆管介导的信号在整个过程中的核心作用。此外,研究发现Atoh8作为一种能够限制肝细胞增殖的调节因子,为肝脏从损伤到修复的转换提供了新的视角。这项研究不仅为理解区域性肝损伤提供了宝贵的数据资源,也为促进胆汁淤积疾病中的肝细胞再生提供了潜在的干预策略。
吴柏华、申屠忻怡、南海涛、郭鹏程和郝世杰为胆汁淤积损伤与再生时空图谱论文共同第一作者,惠利健、Miguel A. Esteban、刘龙奇和赖毅维为该研究的共同通讯作者。
肝脏稳态和切除再生时空图谱
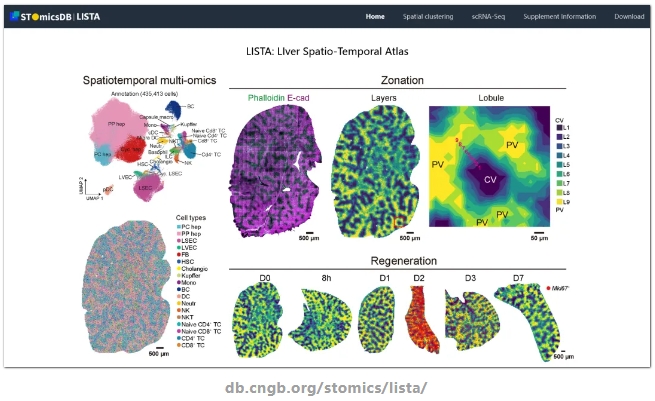
在探索正常稳态和切除再生的研究中,杭州华大生命科学研究院联合吉林大学、中国科学院分子细胞科学卓越创新中心、广州医科大学第五附属医院、山西医科大学、IRCCS-Istituto Tumori、汉诺威医学院等机构,在同期的Nature Genetics杂志上发表了题为A spatiotemporal atlas of mouse liver homeostasis and regeneration的研究论文。这项研究中,研究者以前所未有的空间分辨率揭示了小鼠肝脏在稳态和再生过程中的细胞类型、基因表达和微环境信号的时空动态变化特征。在肝脏再生方面,研究揭示了肝实质细胞和非实质细胞间如何通过分子梯度和基因网络动态互作来促进肝脏的快速恢复,特别是发现了转录共激活因子Tbl1xr1在连接炎症反应与能量代谢,进而促进肝细胞增殖中的关键作用。
研究首先构建了连续的肝小叶网络,发现了肝实质细胞、内皮细胞和肝星状细胞在门静脉-中央静脉轴上的区带化分布特征,并识别出一系列具有区带化分布的基因,如中间区高表达的甲状腺激素受体Thrb和两端高表达的肝细胞增殖抑制因子Bmp4等。此外,研究还发现了肝脏中存在约50~200 μm大小的"immune spot"微结构,丰富了对肝脏免疫微环境的认识。
在经典的2/3部分肝切除模型中,研究阐明了肝再生过程中代谢及细胞间调控网路在空间上的协调机制。研究发现,尽管再生过程中肝脏仍保持分区特征,但各分区的代谢功能均有所下调;不同分区的细胞应答存在差异,如中央静脉区Wnt和NF-κB通路激活更高,门静脉区IL6通路激活更高,中间区核糖体生成更活跃。通过空间基因调控网络分析,研究鉴别了一系列参与肝脏分区维持的重要转录因子,并阐明了基因调控网络在时空上协调肝脏再生的机制。其中,研究发现转录辅因子Tbl1xr1在肝切除后被炎症信号激活,敲降Tbl1xr1可显著抑制肝细胞增殖,证明其在肝再生中的关键调控作用。该研究为正确理解肝脏稳态和再生提供了丰富的数据资源,也为未来绘制哺乳动物肝脏生理和功能障碍的高清晰度时空图谱奠定了基础。
许江山、郭鹏程、郝世杰、上官顺成、石泉和Giacomo Volpe为肝脏稳态-2/3切除-再生时空图谱论文共同第一作者,Miguel A. Esteban、赖毅维、陈奥、刘龙奇、徐讯和郭鹏程为该研究的共同通讯作者。
上述研究得到了国家自然科学基金(集成项目)、科技部、上海市科委等的项目资助,以及深圳国家基因库和广东省基因组科学数据中心的帮助与支持。
胆汁淤积与再生时空转录图谱数据库(CIRSTA)和肝脏时空图谱数据库(LISTA)由CNGBdb团队开发及维护,使用过程中遇到任何问题或有意见建议均可通过以下方式反馈给我们:
使用CNGBdb官网反馈按钮:访问官网(https://db.cngb.org/)→ 点击首页右侧反馈按钮 → 填写意见和邮箱 → 提交
发送意见至邮箱:CNGBdb@cngb.org> 本文留言区留言
原文链接:https://www.nature.com/articles/s41588-024-01687-whttps://www.nature.com/articles/s41588-024-01709-7
信息来源于:“BioArt”公众号
最新文章
- 国家基因库亮相第四届山海连城自然深圳生活节2024-11-11CNGBdb
- “深”生不息 - 国家超算互联网生态沙龙在深圳国家基因库顺利开展2024-11-08CNGBdb
- Cell发表全球首份多器官衰老时空图谱,助力延缓衰老2024-11-07CNGBdb
- The Innovation发表百合高质量染色体级别基因组2024-11-05CNGBdb
- 国家基因库成为“湾区语料联盟”首批成员单位,助推大湾区AI产业高质量发展2024-11-04CNGBdb


