BASS:为单细胞分辨率的空间转录组学提供多尺度和多样本分析
空间转录组学研究正在达到单细胞空间分辨率,数据通常来自多个组织切片。《Genome biology》发表了一种计算方法BASS,支持单细胞分辨率空间转录组学的多尺度和多样本分析。BASS在单细胞尺度上进行细胞类型聚类,在组织区域尺度上进行空间结构域检测,这两项任务在贝叶斯层次结构模型框架内同时进行。

BASS是什么?
目前用于细胞类型聚类和空间结构域检测的方法存在两方面的局限性:1)所有现有方法仅执行两个分析任务中的一个,有效地使两个不同解剖尺度的分析相互分离;2)所有现有方法都集中于分析从单个组织切片收集的空间转录组学数据。基于此,开发团队提出了一种新的计算方法BASS,用于多尺度和多样本分析,克服了上述两个限制。
BASS以联合细胞类型聚类和空间结构域检测的形式进行多尺度转录组学分析,这两项分析任务在贝叶斯分层建模框架内同时执行。对于这两种分析,BASS适当考虑了空间相关性结构,并将基因表达信息与空间定位信息无缝集成,以提高其性能。此外,BASS能够进行多样本分析,联合建模多个组织切片/样本,促进跨组织样本的空间转录组学数据整合。
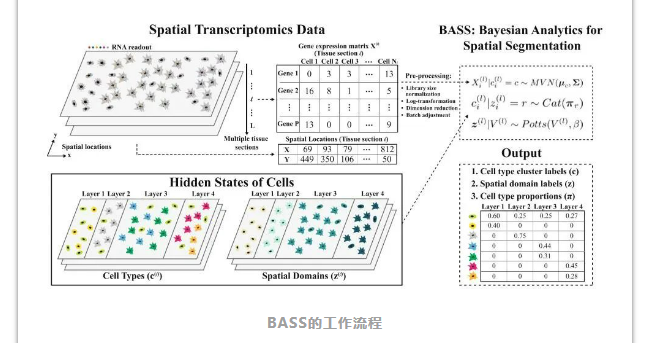
BASS进行多尺度和多样本分析,以便在空间转录组学中进行准确的细胞类型聚类和空间域检测。BASS接受来自多个组织切片的基因表达矩阵和空间位置信息的输入,并在一个分级贝叶斯框架中对这两种信息进行建模。在分析中,BASS将组织上每个细胞的细胞类型标签(c)和空间结构域标签(z)作为潜在/隐藏的变量,并通过一个有效的推理算法推断它们。分析后,BASS提供推断出的细胞类型集群标签、空间结构域标签和每个空间结构域内的细胞类型比例作为输出结果。
BASS的性能测试
开发团队通过对三个数据集的全面模拟和应用来说明BASS准确地揭示大脑皮层和下丘脑的转录组和细胞景观的巨大优势。
模拟数据
首先,开发团队评估了不同方法在单个组织切片上进行空间结构域检测的性能:在模拟中发现BASS在所有场景中都优于HMRF、BayesSpace和SpaGCN。
接下来,评估了不同方法在单个组织切片上细胞类型聚类的性能:在模拟中发现BASS在所有场景中都优于SC3、Seurat和FICT,当细胞类型在组织上显示空间模式时,BASS优于其他三种方法的优势尤其明显。
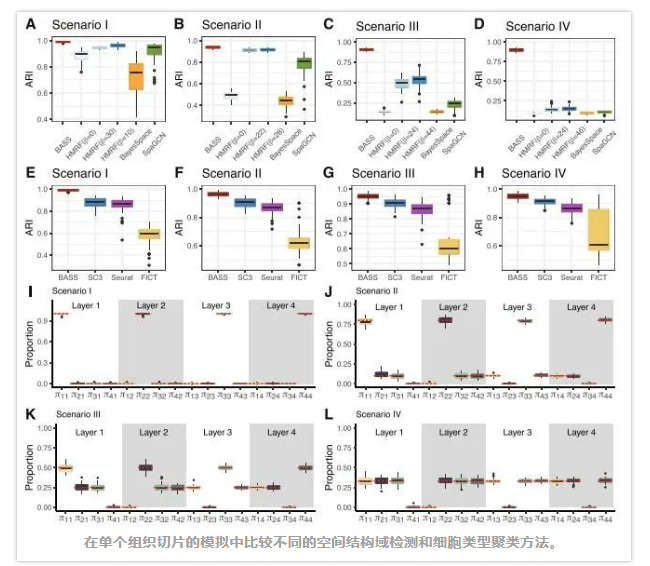
最后评估了BASS在多个组织切片综合分析中的性能:在所有模拟场景中所有三个分析任务(包括空间结构域检测、细胞类型聚类和跨域细胞类型组成估计)的BASS性能随着分析组织切片数量的增加而增加。对于细胞类型聚类,Seurat的性能也随着分析的组织切片数量的增加而增加,而SC3的性能先增加,然后随着组织切片数量增加而降低。与单个组织切片分析类似,当细胞类型在组织上显示出空间模式时,BASS相对于其他方法在细胞类型聚类上的优势尤其明显
除了上述的主要模拟,开发团队还探讨了其他各种因素,包括特定细胞类型/空间结构域的数量、稀有的细胞类型、以及随机排除基因对不同方法性能的影响。
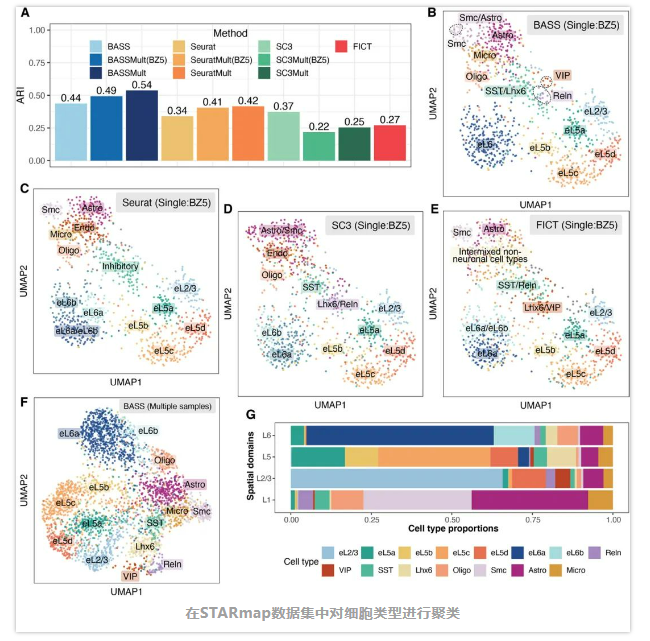
小鼠内侧前额叶皮层的STARmap数据
首先,开发团队检查了不同方法对空间结构域检测的结果:BASS检测到的四个空间结构域与预期的皮质层高度相似(ARI=0.82)。相比之下,HMRF(ARI=0.57)、BayesSpace(ARI=0.26)和SpaGCN(ARI=0.36)检测到的空间结构域与基本事实基本不匹配,方法的排名与模拟数据中观察到的基本一致。此外,BASS检测到的四个皮质层之间有很好的隔离,它们之间有平滑的边界,而BayesSpace和SpaGCN检测到的皮质层在某种程度上交织在一起。值得一提的是,BASS的多样本综合分析能力进一步提高了用于一个样本分析的同一组织切片上的空间结构域检测精度。此外,与我们在焦点样本中发现的情况类似,BASS在单切片和多切片分析中都准确地捕捉到了其他两个切片的四个空间结构域,比其他方法更准确。这些结果证实了BASS在空间结构域检测和多样本综合分析方面的优越性。
接下来检验了不同方法细胞类型聚类的结果:单个组织切片上与模拟一致,BASS实现了准确的细胞类型聚类(ARI=0.44),比Seurat(ARI=0.34)、SC3(ARI=0.37)和FICT(ARI=0.27)更准确;BASS的多样本分析也优于其他两种细胞类型聚类方法(Seurat和SC3)。
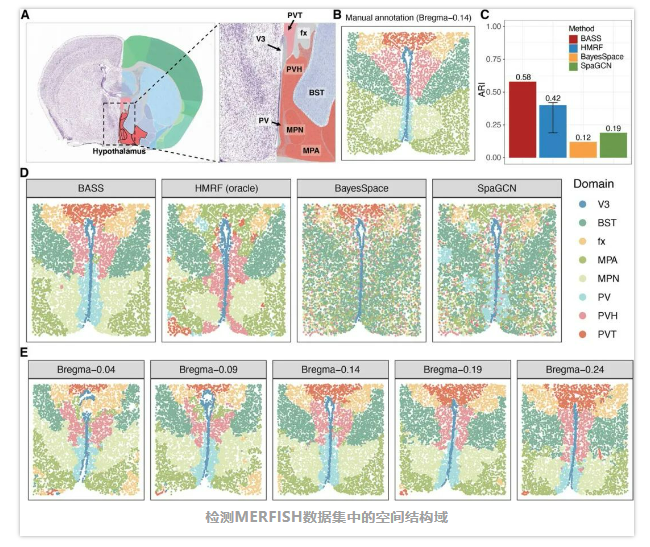
小鼠下丘脑的MERFISH数据
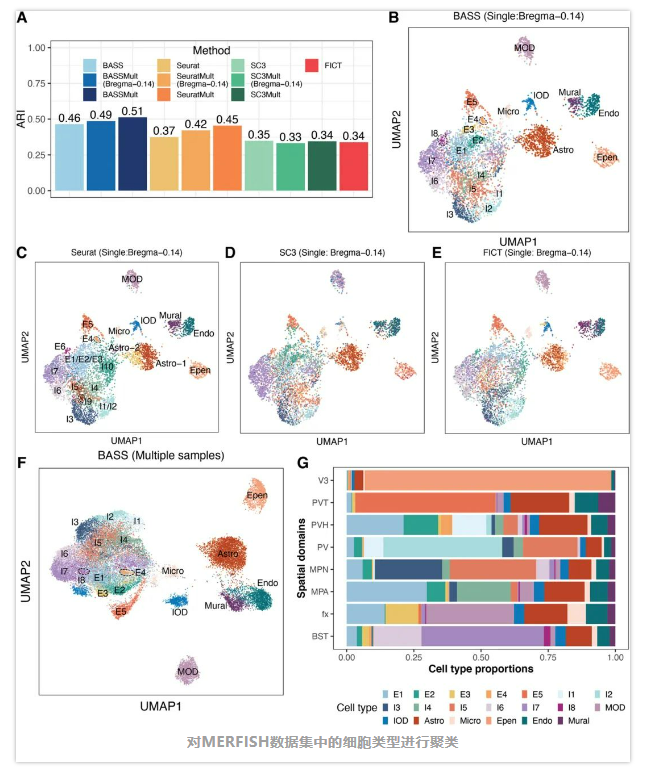
首先,开发团队检查了不同方法对空间结构域检测的结果:BASS检测到与基础组织学注释高度相似的主要空间结构域(ARI=0.58)。相比之下,由HMRF(ARI=0.42)、BayesSpace(ARI=0.12)和SpaGCN(ARI=0.19)检测到的空间结构域通常与基本事实不匹配。重要的是,利用BASS对五个相邻空间转录组组织切片进行多样本综合分析,进一步深入了解了下丘脑视前区的结构组织,这是其他方法的单样本分析无法实现的。
接下来检验了不同方法细胞类型聚类的结果:单个组织切片上与模拟一致,BASS实现了准确的细胞类型聚类(ARI=0.46),比Seurat(ARI=0.37)、SC3(ARI=0.35)和FICT(ARI=0.34)更准确。使用BASS的多样本分析产生了与Bregma-0.14的单个切片分析类似的细胞类型聚类准确度(ARI=0.49)。使用Seurat的多样本分析改进了单个切片分析(ARI=0.42),而使用SC3的多样本研究与单个切片分析相比产生了较低的细胞类型聚类性能,这可能是由于其性能随着细胞数量的增加而降低(如前所述,ARI=0.33)。三种方法的多个切片与单个切片分析的比较与模拟结果一致,突出了BASS的优势。
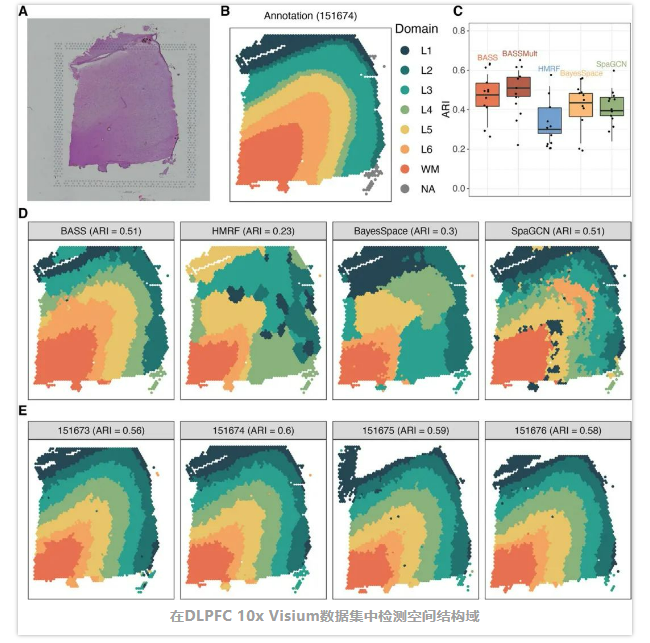
人背外侧前额叶皮层的10x Visium数据
在单个切片分析中,BASS检测到与大多数组织切片的基本组织学注释高度相似的主要空间结构域(跨切片的平均ARI=0.48),比HMRF(平均ARI=0.30)、BayesSpace(平均ARI=0.44)和SpaGCN(平均AR1=0.40)更高。重要的是,对来自每个成人供体的四个组织切片的综合分析进一步改进了空间结构域检测(中值ARI=0.51),并在四个切片上产生了一致的空间结构域。用BASS进行的多样本综合分析大大改善了这个特定样本的空间结构域的检测,并产生了与基础注释高度相似的皮质层,而且在所有四个切片中都是一致的(ARI = 0.60)。在其他两组组织切片中也可以进行类似的观察,证实了BASS在非单细胞分辨率空间转录组学中的空间结构域检测和多样本综合分析的性能。
在运行时间和内存使用方面,BASS与其他空间结构域检测方法和细胞类型聚类方法相当。对于典型的10x Visium数据,大约5000个点,BASS需要大约8分钟,并使用2GB内存。此外,BASS的运行时间和内存使用量与数据的样本大小呈线性关系,这使得BASS可扩展到分析成千上万个细胞/点。
尽管BASS、HMRF和BayesSpace都采用了Potts模型,但BASS在Potts模型的基础上引入了额外的分层建模结构,以便进行灵活和有效的空间转录组建模。与HMRF和BayesSpace相比,额外的分层建模结构使BASS能够对空间结构域的组成做出根本性的不同和更有效的假设。BASS在分层建模结构中引入了细胞类型组成作为中间层,以明确地模拟不同细胞类型的不同基因表达。因此,BASS能够将一个空间结构域定义为具有独特细胞类型组成的区域,以更好地捕捉每个空间结构域内的基因表达异质性,并实现更好的性能。此外,BASS根据现有数据推断Potts模型中的空间相互作用参数β,而HMRF和BayesSpace则将该参数改为用户指定的值。BASS中对β的推断也有助于提高其性能。最后,从模型推理的角度来看,尽管BASS和BayesSpace都是基于贝叶斯框架,但BASS使用Swendsen-Wang算法对空间结构域标签进行采样,而BayesSpace使用Gibbs采样算法。Swendsen-Wang算法比Gibbs采样算法有更好的混合率,因此也有助于BASS实现更好的准确性。
在必要的预处理步骤之后,BASS也可以应用于其他空间转录组学技术,例如HDST和Seq-Scope。
BASS在未来有几个重要的扩展。例如开发团队主要侧重于使用基因表达数据作为输入,但注意到BASS建模框架是灵活的,可以很容易地以额外特征输入的形式纳入组织学图像的信息,以进一步提高其性能。另一个例子是用主成分分析(PCA)中归一化基因表达矩阵的低维成分作为BASS的输入,且BASS并不局限于PCA,它可以与其他降维方法配对,以其低维分量作为输入。
此外,BASS检测到的准确的细胞类型和空间结构域可以与许多其他分析工具配对,以进一步改善各种下游应用,揭示更多的生物学见解,例如与SpatialCorr配对,以检测组织区域内或组织区域之间具有协调调节的基因。
BASS 是作为 R 包实现的, BASS 软件以及用于重现本研究中呈现的所有分析结果的代码可在 GitHub 和 Zenodo 上免费获得:
参考文献
Li Z, Zhou X. BASS: multi-scale and multi-sample analysis enables accurate cell type clustering and spatial domain detection in spatial transcriptomic studies[J]. Genome biology, 2022, 23(1): 1-35.
图片来源于Genome biology官网和参考文献,如有侵权请联系删除。
